- Lunes, marzo 31, 2025
INVESTIGACIÓN MEXICANA AVANZA EN EL ENTENDIMIENTO DE LA NATURALEZA DEL ROTAVIRUS
Academia Mexicana de Ciencias
Boletín AMC/160/16
Ciudad de México, 26 de julio de 2016
- Los avances contribuyen al conocimiento de estrategias terapéuticas que se pueden utilizar en el tratamiento de infecciones virales.
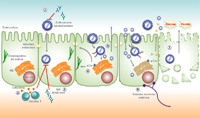
- El rotavirus afecta prácticamente a todos los niños alrededor de los cinco años de edad y es en todo el mundo la principal causa de diarreas deshidratantes en los niños, establece la UNICEF. En la imagen: modelo de entrada de rotavirus en la célula.
ilustración: cortesía de la doctora Daniela Silva Ayala.
Galería de imágenes
La infección por rotavirus es la principal causa de gastroenteritis infantil a nivel mundial, principalmente en países en vías de desarrollo. Se estima que alrededor de los cinco años de edad, prácticamente todos los niños se habrán infectado al menos una vez.
Los rotavirus aprovechan la función de múltiples proteínas hospederas para su propio beneficio durante el proceso de infección, por lo que la viróloga Daniela Silva Ayala decidió desarrollar su tesis doctoral basada en “Identificación de proteínas celulares necesarias en la infección por rotavirus utilizando genómica funcional”, título de su investigación, con la cual se llegaron a importantes resultados.
“Los virus necesitan de la célula para replicarse, por ello nuestro interés fue encontrar y definir aquellas proteínas celulares que ‘ayudan’ al virus, nos preguntamos cuáles eran esos elementos biológicos de la célula hospedera o de la célula infectada que estaban sirviéndole al virus para que este pudiera completar su ciclo replicativo”, indicó.
Esto es relevante para las estrategias terapéuticas que se pueden utilizar en el tratamiento de infecciones virales, para atacar al hospedero o a aquellas proteínas celulares que son las que permiten este tipo de infecciones, así que el trabajo de la investigadora fue identificar qué moléculas de la célula estaban permitiendo que la infección sucediera.
Con su trabajo de tesis, dirigido por el virólogo y miembro de la Academia Mexicana de Ciencias (AMC), Carlos Arias Ortiz, Silva Ayala demostró la participación directa de la maquinaria ESCRT (conjunto de varias proteínas que se encargan de introducir moléculas que se encuentran en el exterior de la célula e internalizarlas mediante un proceso llamado endocitosis) en el ciclo de vida de un virus sin envoltura y abonó en el conocimiento sobre el mecanismo complejo que estos virus utilizan para entrar en las células.
“El virus distrae a la célula haciéndola creer que es una molécula extracelular necesaria y que debe ser internalizada a través de esta maquinaria que se ha descrito en la comunidad científica en los últimos seis años, es una forma en que la célula come lo que está al exterior”, anotó.
Respecto a los virus con o sin envoltura, algunos tienen en su parte exterior una envoltura de lípidos que adquieren cuando salen de su célula hospedera, pero hay otros virus que se hallan desprovistos de envoltura como rotavirus; es decir, que no adquieren esa capa de lípidos en su parte exterior y es así como se definen los virus cubiertos o descubiertos. “Rotavirus y todos los virus de la familia Reoviridae son no envueltos, no tienen esa membrana celular adquirida en su proceso de salida de la célula”.
Daniela Silva Ayala fue reconocida con el Premio Weizmann 2015 en el área de ciencias naturales, el cual otorga la AMC y la Asociación Mexicana de Amigos del Instituto Weizmann de Ciencias por este trabajo de tesis doctoral, realizado en el Programa de Ciencias Bioquímicas del Instituto de Biotecnología de la UNAM.
Cepas de rotavirus estudiadas
Otro de los logros obtenidos con este estudio fue caracterizar detalladamente el tráfico de las cepas UK y RRV de rotavirus en el espacio intracelular. “Estas cepas son las que se encuentran más caracterizadas y son sumamente adaptables a cultivos celulares, por lo tanto, es muy fácil trabajar con ellas, crecerlas y hacer experimentos; son dos que representan muy bien a la población de las diferentes cepas de rotavirus que se encuentran en el mundo, así, los resultados nos hablan de generalidades que comparten con muchas otras cepas”, desatacó.
La aportación más importante que se hace con esta investigación, en opinión de la Silva Ayala, además de identificar posibles blancos para futuros tratamientos terapéuticos, es que habla de la naturaleza intrínseca de una infección viral, se explica cómo la célula ante una infección viral puede cambiar totalmente su estatus biológico, “nos está enseñando la capacidad de un virus de reprogramar toda la célula, las diferentes fases que tiene esta tras una infección viral, es decir, nos enseña sobre la naturaleza del virus y de la propia célula”.
El trabajo en el laboratorio
Aunque para el rotavirus hay una vacuna, la especialista aseguró que esta no es totalmente eficiente: “Desde hace dos años existe una contención de la enfermedad, pero sigue habiendo un gran número de niños que siguen muriendo, por lo que se requiere buscar más alternativas, vacunas y otros tratamientos, sobre todo para niños que ya se encuentran infectados”.
Por lo anterior, señaló que los resultados de las tesis de doctorado son importantes y relevantes porque se habla de posibles terapias, no para prevenir la infección, sino para combatirla.
Para llegar a los resultados citados en el trabajo, la investigadora realizó análisis de silenciamiento de expresión de genes conocido como interferencia de RNA (RNAi) a gran escala, los cuales son eficientes como herramienta de genética reversa (se parte de una región de ADN [gen], que se quiere estudiar y mediante la inhibición de este gen se espera observar un fenotipo, una alteración del individuo [en este caso células en cultivo celular] que ayude a dilucidar cuál es la función de dicho gen, en este tipo de estudios se emplean técnicas de genética molecular para llevar a cabo la inhibición de la función de un gen en particular) para el estudio de la interacción de virus con sus células huésped.
El análisis de los datos obtenidos demostró que las proteínas definidas como positivas se encuentran implicadas en un gran rango de funciones celulares, sugiriendo varias vías metabólicas que pudieran estar relacionadas en el ciclo replicativo del rotavirus.
Así, se realizó un análisis global para identificar factores celulares requeridos en el ciclo replicativo de este virus. Se utilizó un ensayo a gran escala en el que se evaluó el efecto de una biblioteca de siRNAs dirigida contra aproximadamente 23 mil genes celulares, se identificaron 522 genes cuyo silenciamiento por RNAi aumentó o disminuyó la expresión de proteínas virales en células infectadas con rotavi
Las vías celulares más relevantes en este análisis fueron las vías ubiquitina-proteasoma, uniones estrechas y procesos de endocitosis (la célula introduce moléculas grandes o partículas). De particular interés fueron 15 proteínas celulares que participan en procesos de endocitosis y que pudieran participar en la entrada del virus.
En la actualidad, Daniela Silva Ayala realiza un posdoctorado en Harvard Medical Schoool en el Departamento de Microbiología e Inmunología, laboratorio que trabaja específicamente con virus, encabezado por el doctor Max Nibert. Uno de los dos proyectos en los que está enfocada es en relación con la microbiota del estómago; el interés aquí es saber qué tan importantes son ciertos elementos de esta fauna intestinal para combatir infecciones de virus gastrointestinales como rotavirus.
Elizabeth Ruiz Jaimes.
Regresar Arriba, o a Comunicados, o al Inicio.
AMC "Casa Tlalpan" Calle Cipreses s/n, km 23.5 de la carretera federal México - Cuernavaca, San
Andrés Totoltepec, Tlalpan, C.P. 14400, México, D.F.
Coordinación de Comunicación y Divulgación
Teléfonos: (52-55) 58 49 49 04, Fax: (52-55) 58 49 51 10, amcpress@unam.mx
Mapa de ubicación
